Bien choisir les outils que l’on utilise au quotidien est important. Pour ma part, je veux des outils avec lesquels je suis à l’aise, qui soient suffisamment performants et qui produisent des rendus de bonne qualité.
Dans la vie de tous les jours, je suis physicien. Plus précisément, j’étudie la photo-physique de semi-conducteurs un peu exotiques. Concrètement, cela signifie que je passe beaucoup de temps en salle de manipe à collecter des données de spectroscopie. Je ne suis donc pas un physicien théoricien qui utiliserait l’informatique pour faire du calcul symbolique, ou un numéricien qui utiliserait de gros calculateurs pour faire tourner des simulations. Mon utilisation de l’informatique est double :
- Contrôler mon expérience finement;
- Traiter les données acquises, c’est-à-dire réaliser des ajustements de variable sur les données par rapport à des modèles relativement simples, et surtout afficher les données pour alimenter ma réflexion.
Aujourd’hui j’ai choisi de vous détailler un peu la manière dont je réalise la seconde partie. Ceci n’a absolument pas vocation à être une recommandation d’utilisation, ou à dénigrer d’autres manières de travailler. Cependant, si cela peut donner des idées pour s’inspirer, ou si vous pensez que je suis passé à côté d’un outil intéressant, n’hésitez pas à me contacter.
Ce billet a été initialement rédigé pour la newsleter FedeRez de Février 2022 (non disponible publiquement à ma connaissance).
Quels outils existent?
Étant donné la simplicité des tâches que je souhaite réaliser, il existe une foultitude d’outils. Je voulais initialement commencer cet article par une présentation de ceux-ci, mais j’ai eu peur que cela se transforme en une longue diatribe finalement peu productive. Je vous propose donc simplement un gros tableau qui résume les outils dont j’ai connaissance (et je peux râler sur Python, NumPy et Matplotlib en privé).
| Outil | Payant | Libre | Je l’ai utilisé | Plateforme | Divers |
|---|---|---|---|---|---|
| Origin | ✓ | ✗ | ✗ | Logiciel spécialisé | Très utilisé dans le monde de la recherche, il produit de bons rendus, mais le prix d’achat des licences est élevé. |
| Igor | ✓ | ✗ | ✓ | Logiciel spécialisé | L’utilisation n’est pas très intuitive à mon avis. |
| Régressi | ✗ | ✓ | ✓ | Logiciel spécialisé | Très apprécié par les profs de Lycée, mais on est rapidement limité. |
| Microsoft Excel (et clones libres) | ✓ | ✗ | ✓ | Logiciel spécialisé | Les rendus ne sont pas à la hauteur d’une publication scientifique. |
| MATLAB | ✓ | ✗ | ✓ | Langage de programmation | Très utilisé. L’API pour les graphes est rapidement pénible, le prix des licences est élevé et on se trouve rapidement à devoir payer des toolboxes supplémentaires pour des fonctionnalités basiques. |
| Scilab | ✗ | ✓ | ✓ | Langage de programmation | Un clone de MATLAB, les performances en moins. |
| GNU/Octave | ✗ | ✓ | ✓ | Langage de programmation | Un clone de MATLAB, les performances en moins. |
| Python + Matplotlib + NumPy | ✗ | ✓ | ✓ | Langage de programmation | Très utilisé. L’API de Matplotlib est inspirée de celle de MATLAB, avec laquelle j’ai du mal, de même avec les tableaux NumPy. |
| R | ✗ | ✓ | ✗ | Langage de programmation | Très utilisé. |
À titre personnel j’utilise le langage Julia. Il y a plusieurs raisons à cela :
- Libre et gratuit;
- Je préfère l’indexation row-major des tableaux multidimen- sionnels.
- Assez rapide pour les tâches qui se prêtent bien à la com- pilation Just In Time (JIT), même si ça impose quelques contorsions sur d’autres.
Comment on assaisonne Julia?
Éditer et lancer du code
Julia est relativement jeune, et les outils associés évoluent assez vite. La première approche que j’ai pu employer a été d’utiliser une solution de notebooks. J’ai d’abord essayé Jupyter, mais la nécessité d’avoir le serveur en fonctionnement pour pouvoir lire ses fichiers m’a rapidement refroidi. Je me suis ensuite tourné vers Pluto.jl, une bibliothèque en pur Julia qui propose des notebooks dans lesquels l’état interne de la machine correspond toujours aux cellules affichées dans le navigateur. Je pense que cela a de l’intérêt pour l’apprentissage, mais dans mon cas d’usage, où je suis amené à ouvrir et fermer de nouveaux notebooks de nombreuses fois dans la journée, la perte de temps due à la compilation JIT était trop importante.
Ma solution actuelle est plutôt rustique : j’utilise NeoVim avec deux plugins qui me permettent d’envoyer des cellules (délimitées dans mon fichier Julia par des commentaires spéciaux) vers le REPL (Read-eval-print loop) Julia. NeoVim propose une intégration du Language Server Protocol, ce qui permet d’avoir un linter correct dans l’éditeur, et rend cette solution pour mon utilisation personnelle
En pratique, je fais tourner le REPL dans un tmux pour ne pas le relancer de la journée. De cette manière, on peut éviter de perdre du temps au lancement de chaque script à précompiler les bibliothèques les plus utilisées. J’exécute donc tous mes scripts dans le même environnement, charge à moi d’éviter les collisions. Une alternative serait l’utilisation de la bibliothèque DaemonMode.jl.
J’ai parlé de précompilation. La remarque presque immédiate que l’on peut avoir est qu’il suffirait de sauvegarder sur disque tout le code compilé pour gagner du temps. C’est ce que permet PackageCompiler.jl. Grâce à lui on peut créer une sysimage et demander à Julia de la charger au démarrage pour gagner du temps. Exemple avec la bibliothèque de tracé que j’utilise :
Sans la sysimage spécialement préparée :
julia> @time using GLMakie
9.068354 seconds (14.31 M allocations : 980.654 MiB , 5.96% gc time , 10.16% compilation time )
julia> @time lines (0..5 π , x - > sin (5x)/(2+ cos (x)))
61.196285 seconds (119.76 M allocations :
6.479 GiB , 3.58% gc time , 99.55% compilation time )
julia> @time save (" fig.png " , current_figure())
1.005743 seconds (2.90 M allocations : 160.303 MiB , 2.57% gc time , 98.51% compilation time )
GLMakie.Screen(...)
Avec la sysimage :
julia> @time using GLMakie
0.003112 seconds (1.06 k allocations : 70.516 KiB , 117.37% compilation time )
julia> @time lines (0..5 π , x - > sin (5 x )/(2+ cos ( x )))
3.426703 seconds (5.97 M allocations : 298.850 MiB , 11.13% gc time , 95.23% compilation time )
julia> @time save (" fig.png " , current_figure())
0.078950 seconds (37.27 k allocations : 4.044 MiB , 71.64% compilation time )
GLMakie.Screen (...)
On constate assez immédiatement le gain de temps sur le premier appel. Ce dernier déclenche néanmoins toujours des appels au compilateur, les appels suivants aux fonctions de tracés seront donc plus rapides. Je peux partager la « recette » pour obtenir la sysimage et l’utiliser sur demande.
Afficher des données et gérer son projet
Maintenant les outils avec lesquels je fais de la SCIENCE. D’abord, pour la gestion globale de mes projets, j’utilise le très bon DoctorWatson.jl, qui force une arborescence relativement logique et fournit quelques utilitaires pratiques pour nommer ses fichiers de données et les stocker au bon endroit.
En parlant de données, il faut être en mesure de les charger! La plupart de mes données sont stockées au format CSV. J’ai longtemps utilisé CSV.jl par défaut, avant de me rendre compte un beau jour (c’était un mardi) que la bibliothèque standard de Julia fournissait le très bon DelimitedFiles.jl qui bien que beaucoup plus rustique est chargé plus rapidement et fonctionne également plus rapidement.
Une fois ces données chargées, il faut être en mesure d’effectuer des calculs dessus. Dans mon cas il s’agit essentiellement d’ajustements, qui se font bien avec des outils tels que BlackBoxOptim.jl, Optim.jl ou GLM. Pour la partie statistique, elle est assez simple dans mon cas, et je me contente de Statistics.jl de la bibliothèque standard et de StatsBase.jl.
La dernière (et peut-être la plus importante) partie du travail de traitement de données consiste à les tracer de manière à peu près intelligente. Pour ce faire j’ai utilisé pendant un petit moment Plots.jl. L’idée derrière est assez astucieuse, et cette bibliothèque a très probablement participé à l’essor de Julia. Plutôt que de créer une bibliothèque de zéro, Plots.jl propose une interface unifiée (et assez cohérente) pour de nombreux backends. Avec le même code, on est ainsi capable de créer un tracé avec Matplotlib ou en LaTeX avec PGFPlots! Cependant, de par son design Plots.jl est assez lent, et l’interactivité avec les figures n’est pas assurée. De plus sa documentation est parfois un peu légère. J’ai fini par chercher une alternative plus légère. C’est pourquoi j’utilise aujourd’hui Makie.jl, une bibliothèque de tracé intégralement implémentée en Julia, qui propose à ce jour 3 backends : OpenGL, WebGL, Cairo et RadeonProRender (encore expérimental). On peut donc avoir des figures interactives grâce à OpenGL tout en exportant des figures de bonne qualité avec Cairo (mais moins belles qu’avec PGFPlots, certes).
Sans être au niveau de PGFPlotsX.jl, Makie permet tout de même de produire des tracés de qualité convenable1 :
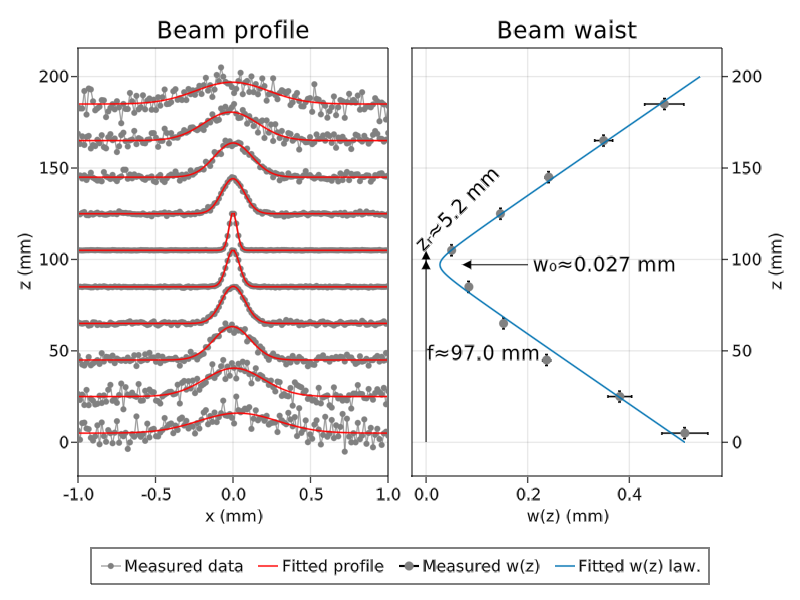
- Si vous êtes un de mes étudiant en TP d’optique, c’est une des figures que j’attends dans les comptes rendus.
↩
Aujourd’hui je pense travailler de manière efficace avec Julia. J’apprécie la syntaxe et la philosophie du langage, et mes outils du quotidien me satisfont globalement. Cependant il y a des points qui peuvent sans nul doute être encore améliorés.
- Le Time to first plot. Malgré la précompilation, il n’est pas rare de devoir attendre une vingtaine de secondes pour afficher mon premier tracé de la journée. L’amélioration des performances de Julia progresse régulièrement, et je suis confiant pour le futur.
- Une plus grande interactivité avec mes figures. J’aimerais être en mesure de déplacer à la souris certains éléments comme les légendes, ou dessiner sur les figures simplement. Actuellement la meilleure option est d’exporter en SVG la figure et de l’ouvrir avec inkscape.
- Plus d’outils de « haut niveau » dans Makie. Par exemple la capacité de numéroter rapidement et simplement les différentes sous-figures d’une figure.
- Un backend de type PGFPlots pour les figures, pour exporter du code LaTeX. J’ai beau chercher, ça reste à mon sens les plus beaux rendus de figure qui soient disponibles, en particulier pour la publication scientifique.


