La médecine actuelle repose en bonne partie sur des médicaments, des molécules, ayant une activité biologique et traitant une maladie. Mais on ne se contente pas de supposer que le principe actif1 du médicament fonctionne, on veut s'assurer qu'il agit correctement et de la manière la plus efficace possible.
Il y a bien naturellement plusieurs façons pour trouver de nouveaux remèdes. Par exemple, certaines plantes sont connues pour avoir des effets pharmacologiques intéressants contre certaines maladies, certains symptômes, avoir des effets apaisants. L'aspirine en est un exemple archi-classique.
Mais nous, on va se concentrer sur la conception ex nihilo d'un médicament pour lutter contre un virus, le VIH (Virus de l'Immunodéficience Humaine). Cela nous servira de fil rouge pour découvrir le cheminement d'un nouveau médicament jusqu'à atterrir dans les pharmacies. Quelles sont les étapes, comment bloquer une infection ? Cela va nous emmener à regarder au niveau de l'infiniment petit, puis de nous servir de ces informations pour élaborer un traitement.
-
C'est, dans la pilule à avaler, la molécule d'intérêt qui va agir. Elle peut être mélangée avec d'autres produits pour faciliter son absorption, voire sa pénétration dans les cellules. ↩
- Préliminaire : connaître les mécanismes de l'infection
- Zoomer sur une molécule : à l'échelle des protéines
- Dernière étape : mettre enfin au point le médicament !
Préliminaire : connaître les mécanismes de l'infection
Pour savoir comment agir, il faut déjà savoir sur quoi agir. Nous allons prendre l'exemple du VIH ; on ne le présente plus. Enfin si, on va le présenter quand même, histoire de bien être sûr de maîtriser ce dont on parle.
Les virus sont des particules biologiques qualifiés de parasites cellulaires obligatoires. Cela signifie qu'ils vont parasiter une cellule hôte en entrant à l'intérieur et en la forçant à effectuer des tâches biologiques qu'elle n'aurait pas faites autrement. Le virus reprogramme génétiquement la cellule à son avantage. Cette reprogrammation cellulaire va ainsi lui permettre de se multiplier et de se propager. Le virus ne peut pas se multiplier seul : il a impérativement besoin d'infecter une cellule.
Pour cela, le virus a besoin :
- d'une information dite information génétique, qui lui permettra de savoir comment se répliquer à l'identique et dont le support est soit l'ADN soit l'ARN,
- d'une couche protectrice faite de protéines appelée capside qui englobe le support génétique,
- de quoi interagir avec une cellule pour s'y introduire ou au moins injecter son information génétique et quelques autres protéines nécessaires à l'expression de cette information génétique.
C'est le minimum syndical du virus. Bien entendu, il peut avoir d'autres choses supplémentaires.
On vient de parler d'ADN et d'ARN. Grosso modo, l'ADN, ou acide désoxyribonucléique, chez vous, est la molécule de support de l'information génétique. C'est un peu comme un mot : un assemblage de lettres les unes à la suite des autres, de façon ordonnée. En français on a le choix parmi 26 lettres, en ADN parmi 4 : les fameuses bases azotées Adénine, Guanine, Thymine, Cytosine (abrégées A, T, G, C), reliées à des sucres et des phosphates.
Ces bases sont placées les unes à la suite des autres et forment ainsi une chaîne, orientée. On lit l'ADN dans un sens. En français les mots sont lus de gauche à droite et sur la molécule d'ADN c'est la structure de la chaîne qui va définir le sens de lecture. On vient donc de définir un enchaînement de lettres et un sens de lecture : on vient de créer un langage. L'enchaînement des bases azotées définit une information, une séquence. C'est cette information que l'on appelle information génétique De plus, l'ADN est constitué de deux chaînes (ou deux brins) en parallèle qui se font face et qui portent la même information1 ; il est dit bicaténaire.
L'ARN, ou acide ribonucléique, est un peu différent :
- un de ses constituants est le ribose, qui lui donne d'ailleurs son nom (« ribonucléique ») alors que l'ADN utilise à la place un autre sucre, le désoxyribose2,
- il utilise la base azotée U (Uracile) à la place de la base T,
- il n'est constitué que d'une seule chaîne. Il est dit monocaténaire.
C'est pour cela que les noms de ces deux supports se ressemblent ; deux types de molécules très similaires mais différents. L'ARN, lui, grosso modo, est une copie transitoire d'une partie de l'ADN qui va servir à l'expression de l'information génétique.
Cette information génétique sert à la synthèse de protéines, une famille de molécules biologiques. Des molécules qui vont fortement attirer notre attention. L'information génétique sert de patrons à suivre pour recréer les protéines qui serviront à la cellule, ou au virus pour détourner la machinerie cellulaire.
Les protéines sont des molécules biologiques capables de réaliser une ou plusieurs actions biologiques. Par exemple, l'amylase est capable de couper les sucres lents (amidon) en petits éléments pouvant être récupérés par l'intestin et ultérieurement utilisés par l'organisme. L'hémoglobine transporte le dioxygène des poumons aux tissus. Le collagène confère des propriétés d'élasticité aux tissus (et vous le connaissez pour l'élasticité de la peau). L'insuline agit contre l'hyperglycémie en transmettant aux cellules un message, dont la conséquence est la diminution de la glycémie. La liste est longue. On reviendra ultérieurement sur les protéines pour voir comment elles peuvent effectuer ces actions biologiques.
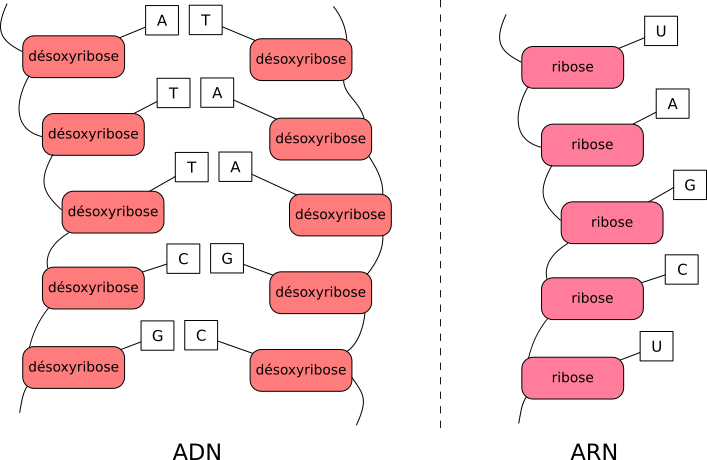
Les bases azotées et le sucre de l'ADN et l'ARN sont légèrement différents.
Avec les mains : l'accès à l'ADN n'est pas chose aisée, alors qu'il faut produire un grand, voire très grand nombre de protéines. Soit vous accédez autant de fois que nécessaire à l'ADN, soit vous faîtes comme quand vous allez à la bibliothèque pour travailler : vous y allez une fois, vous recopiez les passages des livres qui vous sont utiles et vous revenez chez vous avec vos notes. Ce sont ces notes que vous consulterez ensuite, autant de fois que vous en avez besoin. L'ARN, c'est un peu le même principe. Vous accédez à l'ADN un nombre restreint de fois, vous recopiez certains passages d'intérêt et ce sont avec ces copies que vous travaillez. De plus, l'ARN porte une signature chimique différente de celle de l'ADN, donc pas de soucis pour savoir qui est le livre original qui contient tout et qui est le petit pense-bête sur lequel on a écrit uniquement certains passages.
Revenons au VIH. C'est ce qu'on appelle un rétrovirus enveloppé. Rétrovirus parce que son support génétique est sous la forme d'ARN, qu'il va convertir en ADN une fois rentré dans la cellule et l'insérer ensuite dans le génome (c'est-à-dire l'information génétique) de l'hôte. On va reprendre ce point un peu plus loin. Et enveloppé, parce que la capside virale, dans laquelle se trouve l'information génétique et les protéines virales, est elle-même englobée dans une enveloppe (faite de lipides3 et de protéines).
Je case pas mal de jargon de la biologie : génome, parasite intracellulaire obligatoire, brin, lipides, rétrovirus enveloppé, etc. Il est possible que ce vocabulaire vous paraisse très technique. Le but de ces paragraphes n'est pas de retenir tous ces mots d'un coup, mais d'avoir un aperçu de leur signification.
Voici le mécanisme d'action du VIH :

Image Wikipédia - Par Sano
La première étape est l'interaction entre le VIH et la cellule cible. Pour cela, le virus utilise des protéines d'enveloppe gp120 et gp414 (voir l'image suivante qui se focalise sur la particule virale), en interaction avec les récepteurs cellulaires présents à la membrane de la cellule qui sont également de nature protéique.
Les protéines d'enveloppe gp120 et gp41 sont ancrées dans l'enveloppe lipidique du virus. D'abord gp120 interagit avec les récepteurs cellulaires appelés CD4, puis gp41 est alors débloqué et peut interagir à son tour. Ça fait beaucoup de noms mais c'est le principe qui importe. Le virus peut ensuite effectuer une fusion avec la cellule et libérer ainsi son contenu (son information génétique avec des protéines essentielles à l'infection).
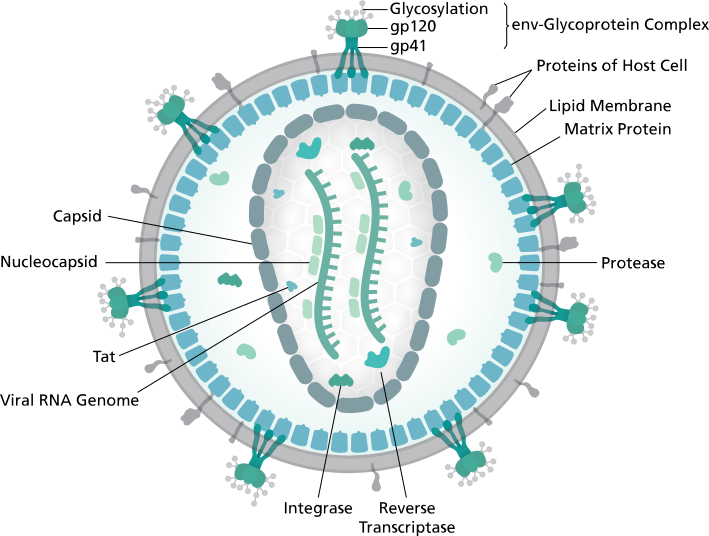
Structure du VIH / wikipédia HIV ; Thomas Splettstoesser (www.scistyle.com)
On retrouve l'enveloppe lipidique avec des protéines d'enveloppe ancrées dedans. À l'intérieur de l'enveloppe, une capside qui referme 2 brins d'ARN, le génome viral, avec quelques autres protéines.
Les protéines d'enveloppe gp120 et gp41 sont impliquées dans les interactions avec les récepteurs cellulaires CD4, nécessaires pour infecter les cellules. Si la cellule ne possède pas de CD4 (ou une particule virale se retrouve avec des gp120 et gp41 non fonctionnelles), l'infection n'aura pas lieu.
On a dit que le VIH est un rétrovirus : son génome a besoin de s'intégrer dans celui de l'hôte pour s'exprimer et donc se multiplier. Pour le VIH, ce n'est pas négociable ; il lui faut insérer son génome dans celui de son hôte, sinon ça ne marchera pas. On a aussi dit que la particule virale qui infecte la cellule a une information sous la forme d'ARN et la cellule hôte sous forme d'ADN. Le VIH est incapable alors d'exprimer son information génétique directement ni de l'insérer dans l'ADN de l'hôte tant que le sien est sous forme d'ARN. Il faut impérativement convertir cet ARN en ADN ; cette étape est appelée rétrotranscription ou transcription inverse.
Ensuite, il faudra intégrer ce bout d'ADN fraîchement produit dans le génome hôte. Pour cela, la particule virale a tout ce qu'il lui faut : une transcriptase inverse (ou reverse transcriptase en anglais), qui convertit l'ARN en ADN et une intégrase, qui insère la nouvelle molécule d'ADN dans le génome hôte. Une fois l'intégration faite, la cellule exprime le génome viral comme si c'était le sien. La cellule produit alors toutes les protéines virales et ainsi reproduit le virus en plusieurs centaines ou plusieurs milliers d'exemplaires. Il a détourné la machinerie cellulaire à son avantage.
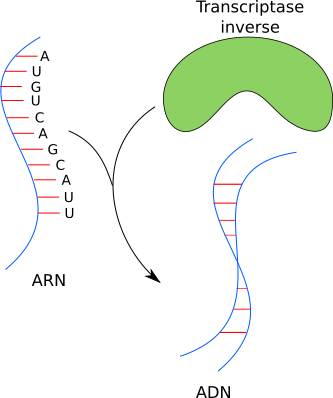
La transcriptase inverse transforme l'ARN en ADN.
Sur cette image, la transcriptase inverse reconnaît l'ARN et crée une copie de cet ARN en ADN. Elle a effectué une action, après reconnaissance de ce qu'on appelle son substrat (ce qu'elle lie, dans notre cas l'ARN), effectue son travail et libère un produit (la copie ADN).
À la fin de son cycle, le virus est produit en masse par la cellule, le VIH bourgeonne des cellules ; les particules virales fraîchement produites se séparent de la cellule et sont libérés. Le cycle viral est terminé. Chaque nouvelle particule virale peut alors infecter une autre cellule.
Physiologiquement, le VIH infecte les cellules dites CD4+, c'est-à-dire celles qui expriment les récepteurs CD4. Il s'agit des lymphocytes T4 et des macrophages, des cellules impliquées dans le système immunitaire.
Les lymphocytes T4 sont appelés T auxiliaires car ils facilitent la mise en place de la réponse immunitaire adaptative, une réponse lente à mettre en place et spécifique d'une infection donnée mais très efficace. Les macrophages sont des cellules immunitaires résidant dans les tissus et participent à la réponse immunitaire innée, une réponse rapide et prenant en charge une multitude d'infections mais moins efficace.
Cette infection par le VIH fragilise le système immunitaire. En effet, lorsqu'une cellule infectée libère de nouvelles particules, cela s'accompagne d'une explosion de la cellule hôte. Par ailleurs, cela amène également le système immunitaire à se combattre lui-même (il est bien conçu pour lutter contre les cellules infectées, non ?). Finalement, cette fragilisation du système immunitaire aboutit au syndrome d'immunodéficience acquise, le SIDA.
Toujours plus dans le détail, le VIH a recours à d'autres protéines accessoires, qui jouent quelques rôles à différents moments du cycle viral. La protéine transactivatrice Tat, la protéine Vif, entre autres.
Toutes les protéines du VIH / image wiképdia - Thomas Splettstoesser
Cette image illustre les protéines codées par le VIH. Grosso modo, on retrouve dedans les protéines d'enveloppe gp120 et gp41, la reverse transcriptase RT et des protéines de capside (celles du regroupement « gag ») avec quelques autres protéines, qu'on ne détaillera pas. Retenons simplement que le VIH a recours à chacune d'entre elles à un moment de son cycle réplicatif. En conséquence, chacune mérite qu'on s'y penche un peu. Certaines protéines sont peut-être plus utiles que d'autres donc plus intéressantes pour nous à analyser, il n'empêche qu'inactiver chacune mettrait des bâtons dans les roues du virus.
Bien entendu, chaque découverte nécessite des mois de travail et personne ne travaille sur l'ensemble de ces acteurs. En revanche, chacune de ces étapes est un point-clef pour empêcher le VIH d'accomplir son cycle viral. On peut empêcher les interactions entre les récepteurs et les protéines d'enveloppe du VIH ; on peut empêcher la rétrotranscription (exemple d'un autre traitement : l'azidothymidine) ; on peut inhiber l'action de l'intégrase ; on peut bloquer les protéines accessoires pour bien pourrir la vie au virus ; on peut bloquer le départ des particules virales de la cellule et donc empêcher sa libération complète.
Enfin, tout ça c'est de la théorie.  Il manque à passer à la pratique. Cette connaissance préalable nous donne des acteurs potentiels sur lesquels intervenir. Une étude du terrain, en somme. C'est autant d'informations utiles pour nous. Récapitulons : intervention des protéines gp120 et gp41 pour la fusion avec la cellule (on va en rediscuter plus tard), intervention de la transcriptase inverse pour transformer l'ARN en ADN et une intégrase virale pour injecter le génome au bon endroit. Avec des protéines accessoires, dont on n'a pas trop parlé, mais qui peuvent toujours être intéressantes à cibler. Après tout, elles fournissent un avantage au virus pour bien reprogrammer la cellule ; en la ciblant on ennuie fortement le cycle réplicatif du virus, non ? Bref, cela correspond à autant de cibles possibles.
Il manque à passer à la pratique. Cette connaissance préalable nous donne des acteurs potentiels sur lesquels intervenir. Une étude du terrain, en somme. C'est autant d'informations utiles pour nous. Récapitulons : intervention des protéines gp120 et gp41 pour la fusion avec la cellule (on va en rediscuter plus tard), intervention de la transcriptase inverse pour transformer l'ARN en ADN et une intégrase virale pour injecter le génome au bon endroit. Avec des protéines accessoires, dont on n'a pas trop parlé, mais qui peuvent toujours être intéressantes à cibler. Après tout, elles fournissent un avantage au virus pour bien reprogrammer la cellule ; en la ciblant on ennuie fortement le cycle réplicatif du virus, non ? Bref, cela correspond à autant de cibles possibles.
-
À discuter, en vrai. Les chaînes sont dites en antiparallèle car elles sont placées dans des sens de lecture opposés. En outre, les bases azotées qui se font face ne sont pas identiques. En face d'un A on trouvera un T et réciproquement, en face d'un G on trouvera un C et réciproquement. Cette complémentarité de base permet de retrouver la séquence de l'autre brin, mais les deux brins ne portent exactement la même information. ↩
-
Les noms très proches suggèrent que ces deux sucres sont finalement assez similaires. Ils le sont : on passe de l'un à l'autre par réaction d'oxydoréduction. Cette petite modification est suffisante pour que la cellule les distingue assez bien. ↩
-
Ils sont davantage connus sous le nom de graisses. Il existe de nombreuses familles de lipides, dont le point connu est leurs propriétés de ne pas se mélanger (ou alors très mal) avec l'eau. ↩
-
Ces noms improbables sont donnés en rapport avec leur propriétés. GP pour glycosylated protein ou protéine glycosylée, c'est-à-dire à laquelle la cellule a rajouté certains glucides par-dessus et 120 par rapport à son poids moléculaire, exprimé en kilodaltons (voir dalton). Comme il existe un nombre infiniment grand de protéines, certaines sont nommées sommairement. ↩
Zoomer sur une molécule : à l'échelle des protéines
Avec les études du terrain préalable, on a une vue d'ensemble des acteurs. Maintenant on veut pourrir la vie de chacun de ces acteurs et inhiber chaque étape ou au moins la perturber le plus possible. Pour ça, il faut zoomer davantage sur chaque protéine ; la simple connaissance de sa fonction n'est pas suffisante pour ce qu'on veut faire.
Première chose : repenchons-nous sur les protéines.
Les protéines, des agents biologiques
Par définition, une protéine est une succession d'acides aminés, qui s'assemble ensuite en une unité fonctionnelle.
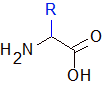
Structure d'un acide aminé seul.
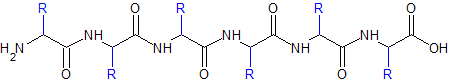
Les acides aminés s'assemblent en un polymère de façon linéaire.
Les acides aminés ont tous une structure commune sur laquelle vient se greffer un radical. Une fonction amine et une fonction acide qui servent à faire des ponts entre les acides aminés, et entre les deux, un carbone qui porte un radical (noté R sur la figure) ou chaîne latérale. C'est le radical qui va différencier un acide aminé d'un autre et lui conférer des propriétés intéressantes : des charges électriques + ou -, des groupements hydrophobes (qui vont fuir l'eau et se débrouiller pour minimiser les contacts avec), etc. Ces groupements induisent des comportements vis-à-vis de l'eau et des comportements entre deux acides aminés et vont participer à structurer la protéine et déterminer son repliement.
Une petite protéine.
La forme globale est donnée par la position de ce qu'on peut désigner comme le squelette acide aminé, soit l'enchaînement des atomes C et N communs à tous les acides aminés (le groupement amine, le carbone central, le carbone acide). Les chaînes latérales dépassent et s'orientent dans l'espace.
Les protéines sont de petite taille - en tout cas comparativement à la taille humaine. Elles sont comprises entre 1 et 10 nm environ. C'est tout petit !
On avait dit également auparavant que les protéines sont des molécules biologiques capables d'effectuer des tâches, ce qu'on n'a pas encore expliqué pour l'instant. On a vu que les protéines peuvent avoir des formes particulières, directement liées à la succession des acides aminés qui les composent. Ce repliement crée des micro-environnements capables d'effectuer une action (lier un substrat ; catalyser une réaction chimique). Faisons une comparaison rapide pour illustrer ce point.
Un moteur peut tourner parce que sa structure lui permet de tourner ; les pistons permettent de récupérer l'énergie libérée par la combustion et d'entraîner la structure, les bougies brûlent le carburant, la pompe d'injection met le carburant. L'assemblage des différentes parties de la voiture, moteur, roues, réservoir de carburant, habitacle, volant et pédales, etc. Bien entendu, une voiture est un assemblage d'un très grand nombre de pièces et à notre échelle. Mais toute proportion gardée, une protéine c'est le même principe, à l'échelle moléculaire. L'intégrase prend en charge l'ADN, reconnaît l'ADN cible, puis insère. Le complexe gp120-CD4 permet l'insertion.
Une protéine va donc avoir plusieurs micro-environnements réalisant chacun une action simple et la réunion de ces petites actions simples fait la fonction. C'est bien la combinaison des acides aminés qui va déterminer les propriétés de la protéine.
On peut aussi mentionner le fait qu'elles peuvent opérer seules ou avoir besoin de s'associer à plusieurs, formant ainsi un complexe. Un complexe fait très souvent apparaître des propriétés nouvelles, de sorte que l'action du complexe est plus que la simple superposition des actions des monomères. La formation du complexe peut faire apparaître des sites actifs que les monomères seuls ne possèdent pas. Cette notion est importante. Si on empêche la formation des complexes, on perd des propriétés et on peut ainsi bloquer l'action.
Encore une fois, la formation d'un complexe ne se fait pas au hasard, mais de façon ordonnée. Reprenons l'exemple de la voiture. Les roues de la voiture ne sont pas placées n'importe où et n'importe comment mais selon un motif précis ; le moteur n'est pas relié au volant mais bien aux roues ni les pédales de frein placées dans le coffre. Les complexes c'est pareil. Il y a un site d'interaction entre les différents acteurs du complexe. C'est au niveau de ce site d'interaction que les éléments élémentaires du complexe, les monomères, viennent interagir et pas ailleurs. La structure est bien ordonnée.

Les deux protéines s'assemblent en une unité fonctionnelle.
Sur cet exemple schématisé, une protéine isolée (en vert, la protéine B) peut reconnaître une molécule mais ne va rien en faire. Une autre (en jaune, la protéine A) peut réaliser une action mais n'a rien pour bien reconnaître sa cible. Elle serait donc peu efficace. Les deux peuvent s'assembler en une nouvelle unité biologique, dotée et d'un site de reconnaissance et d'un site catalytique (réalisant une action chimique).
Pourquoi toute cette explication sur la forme des protéines ? On a dit que la forme permet la fonction. Donc, pour bloquer la fonction, on peut essayer de changer la forme. Les interactions que les radicaux ont les uns envers les autres et avec l'eau (ou avec un substrat) stabilisent la forme de la protéine ; une fixation d'un substrat peut donc stabiliser une autre forme et induire un changement de la structure. Comme on peut s'y attendre, un changement de structure aboutit fréquemment à un changement de fonction.
En conséquence de tout ceci, si jamais on arrive à fixer sur une protéine un petit quelque chose qui perturbe sa fonction en gênant son repliement ou empêchant la formation d'un complexe, on peut bloquer sa fonction. La protéine se retrouve alors avec un boulet aux pieds qui l'empêche de bosser correctement. On parle d'inhibiteurs.
Il faut connaître la structure atomique des protéines
Maintenant, on en sait un peu plus sur les protéines. Revenons à nos moutons : pourrissons la vie du VIH. On a la liste de ses protéines et leur rôle ; on sait ce qu'elles font, mais on ne sait pas comment elles le font. On se doute qu'elles doivent avoir des domaines de reconnaissance, des domaines catalytiques (ce qui effectue une action chimique), mais en fait, là maintenant, on n'a aucune idée de ce à quoi ils ressemblent et de comment ils fonctionnent.
On a besoin de comprendre comment les facteurs cellulaires et viraux interagissent (quelles parties de la protéine d'enveloppe gp120 entrent en contact avec les récepteurs CD4 ; quelles parties de l'intégrase viennent prendre en charge l'ADN viral et l'ADN hôte), on a besoin de savoir comment la rétrotranscription se passe.
Pour cela, on revient sur ce qu'on a dit juste plus haut. La structure des protéines (ou des complexes protéine-protéine) permet d'expliquer la fonction. On a également évoqué le fait que mettre un boulet aux pieds des protéines virales serait une bonne idée pour lutter contre l'infection. On a besoin de connaître la structure des protéines ainsi que la structure du complexe. De la même façon que le marteau n'est d'aucune aide pour visser, de la même façon n'importe quelle molécule ne permettra pas d'inhiber une étape clef de l'infection. Il faudrait des photos très précises de la structure des complexes.
Obtenir une structure moyenne résolution
On pourrait penser qu'il suffit d'un microscope assez puissant pour voir les atomes. Après tout, un microscope permet bien de voir des bactéries, ce qu'on ne peut pas faire à l’œil nu. Peut-on obtenir alors, plus ou moins facilement, la structure atomique de nos petites molécules ?
Réponse : les lois de l'optique nous empêchent de le faire. Si l'objet que l'on veut regarder est de l'ordre de grandeur de la longueur d'onde de la lumière utilisée pour observer, alors il se produit un phénomène de diffraction. L'information peut être considérée comme perdue. Nous avons donc un souci de taille : la lumière qu'on utilise est de l'ordre de la centaine de nm, les protéines de la dizaine nm et les atomes du dixième de nm.

D'après deux images Wikipédia (Fentes d'Young) libres de droit.
Ces images illustrent le principe de la diffraction. À gauche, la lumière issue de l'échantillon traverse des fentes. Ces fentes sont de l'ordre de grandeur de la longueur d'onde de la lumière. Il se produit un phénomène de diffraction. Lorsque la lumière passe à travers ces fentes, chacune se comporte alors comme une nouvelle source de lumière. Les lois de l'optique nous disent alors qu'entre les deux sources vont apparaître des interférences, ce qui est symbolisé par les superpositions de raies. Ces interférences font apparaître des taches de diffraction (les raies de lumière espacées de zones sombres sur l'image de droite). Je vous laisse en apprendre plus par vous-même sur le phénomène de diffraction. Ce qui nous importe ici, c'est que ces phénomènes de diffraction vont limiter la taille de ce qu'on peut voir en microscopie optique. Les petites objets de taille comparable à celles des longueurs d'onde du visible (environ de 450 nm à 700 nm) induisent des diffraction. Les images à de tels grossissement seraient inexploitables.
Il faut donc autre chose, des nouvelles techniques d'observation. Il vous vient peut-être à l'esprit la microscopie électronique. La microscopie électronique à transmission consiste à faire passer un faisceau d'électrons sur l'échantillon à analyser. Pour simplifier, les atomes rencontrés sur le passage absorbent ou dévient les faisceaux d'électrons. Le détecteur d'électrons récupère ensuite l'empreinte des électrons passés à travers l'objet, compte en chaque point l'intensité électronique passée à travers et permet d'obtenir les clichés. On parle de microscopie électronique à transmission.
La molécule est figée à très basse température (elle ne bouge donc plus et elle est un peu plus protégée des dégâts induits par le faisceau d'électrons, qui abîment l'échantillon). Elle est observée sous différents angles ; un ordinateur analyse les différents résultats, fait ses petits calculs et propose une structure de l'objet analysé. Un petit lien (en anglais) pour en apprendre plus sur la cryo-électro-microscopie.
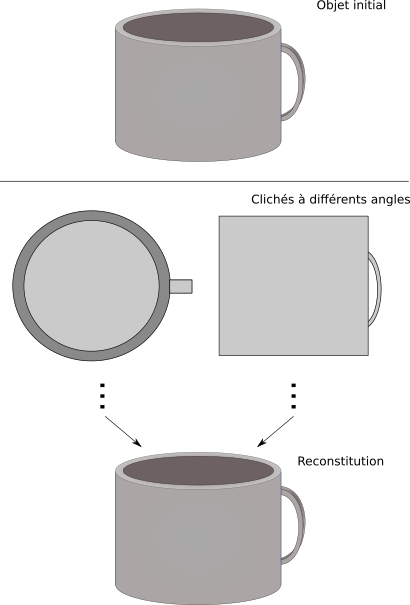
L'objet est observé à différents angles. Les clichés permettent de reconstruire.
Problème : très souvent, ces images ont une résolution assez mauvaise. La résolution se définit, globalement, par la plus petite distance entre deux objets qui peut être distingué sur l'image. En d'autres termes, si deux éléments sont séparés par une distance inférieure à la résolution, on ne pourra pas les distinguer. Au regard des tailles qu'on manipule, on compte très souvent en angström, (Å), avec 1 Å = 10-10 m = 0.1 nm. La résolution va dépendre de la protéine regardée, de la qualité de l'expérimentation, de la précision des détecteurs, de la puissance du faisceau d'électrons, parmi d'autres paramètres.
Globalement, une protéine est de l'ordre de quelques nanomètres (donc de la dizaine à la centaine d’angström). Pour voir le repliement global, on peut considérer qu'il faut une résolution d'environ 10 Å au moins. Enfin, pour voir la position de la plupart des atomes, il faudrait environ 4 Å au moins (1 Å pour voir tous les atomes, notamment l'atome d'hydrogène).
Il arrive que des observations en cryo-électro-microscopie soient suffisamment précises pour avoir la structure atomique. La cryo-électro-microscopie a connu des améliorations fulgurantes et permet de plus de plus d'avoir la structure atomique. Ces améliorations de technique cependant encore peu répandues car très récentes et on aura généralement une résolution de l'ordre de la dizaine d’angström.
Voici un exemple d'une protéine du VIH vue à moyenne résolution en cryo-électromicroscopie :

Une protéine de capside du VIH (entre l'enveloppe et le matériel génétique).
La capside est un assemblage de protéines, situé en dessous de l'enveloppe du VIH.
On observe alors ce qui peut être vu comme étant la surface de la protéine, dans laquelle on peut essayer de positionner certains atomes, ceux du squelette acide aminé. On peut placer dans la structure observée le squelette acide aminé parce que sa forme est contrainte par la linéarité du squelette et par l'enchaînement N - C - C. Cette contrainte permet de placer le squelette dans l'empreinte de la molécule, à peu près correctement. En revanche, les radicaux ont une forme beaucoup plus libre avec plus de possibilités de mouvements. On devrait essayer de placer quelques petits trucs dans un énorme volume, avec aucune assurance qu'on fait le bon choix. On ne fait donc pas figurer les chaînes latérales, par manque d'information. On a une idée de la forme de la protéine mais pas des positions des chaînes latérales.
Obtenir une structure à basse résolution
On est très difficile. On veut plus. Toujours plus. Plus de détail. On en raffole. Il faut voir plus de choses. La structure globale de la protéine est une étape très puissante, mais ce qu'on veut c'est savoir où vont se placer les chaînes latérales ensuite. Pourquoi ? Parce qu'on veut que notre inhibiteur ait une affinité la plus forte possible pour la protéine virale et donc établisse le plus de contacts, les plus précis, les plus fins possibles.
On va donc avoir recours à des méthodes donnant des images plus fines. On va présenter deux méthodes permettant d'arriver à nos fins.
La cristallographie - rayons X
Les deux sont indissociables. Les rayons X, c'est ce qui va permettre de voir. Mais ils ne sont efficaces que sur des cristaux de protéines. Ce qui nous amène à définir la notion de cristal.
Un cristal, c'est une sous-unité qui se répète de façon périodique et très ordonnée. En bref, une unité de base, l'unité asymétrique, qui se répète par translation à l'identique.
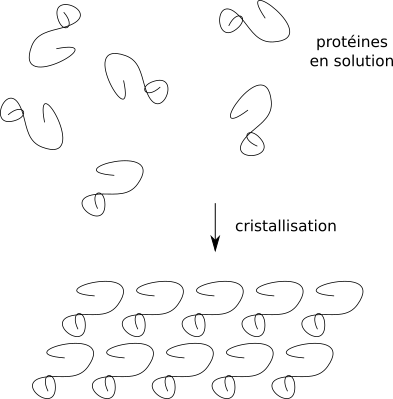
Les protéines s'arrangent en un réseau par répétition d'unité de base.
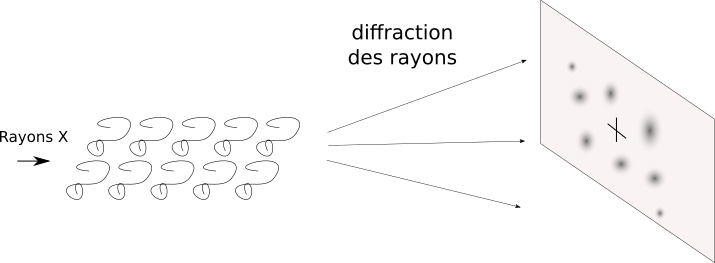
On envoie des rayons X sur une molécule. Les rayons X ont une longueur d'onde comparable à la taille des atomes. Souvenez-vous, la diffraction, les interférences. C'est ce qui va se passer. Ici, la diffraction est ce qui nous intéresse (contrairement à la microscopie optique où elle nous gênait), parce qu'on va analyser les taches d'interférence.
Un faisceau de rayons X qui passe à travers une protéine isolée : rien de bien intéressant ne va se passer. Chaque atome diffracte un peu mais il faut être plusieurs pour faire des interférences. Ce n'est donc pas suffisant pour obtenir un signal. En revanche si les sous-unités se répètent, alors chaque répétition vient ajouter son effet sur le faisceau de rayons X et on pourra avoir des phénomènes d'interférence ; des taches de diffraction vont apparaître.
Les rayons X interagissent avec les électrons des atomes du cristal et sont déviés par phénomène de diffraction. Les taches de diffraction obtenues sont alors caractéristiques de la densité électronique rencontrée par le faisceau.
Plus on s'éloigne du centre du détecteur, plus les intensités sont faibles (la majorité des rayons X sont peu déviés). Or, plus on voit de taches et plus on voit des détails dans la structure, donc plus la résolution sera basse. L'idéal est donc de détecter le plus de taches possible. Cela implique qu'il faut détecter des taches de plus en plus éloignées du centre donc de plus en plus faibles aussi. Pour cela, il faut un cristal le plus gros possible (pour amplifier les phénomènes de diffraction et avoir le plus d'interférence possible) et on peut avoir recours à des sources de rayons X extrêmement puissantes pour augmenter l'intensité du signal.
Les électrons vont dévier les faisceaux de rayons X.
L'image représente de façon simplifiée (certaines propriétés de symétrie ne sont pas représentées) les taches de diffraction. Le détecteur compte le nombre de photons1 qu'il reçoit en chaque point. Les taches de diffraction peuvent ensuite être traitées pour dresser ce qu'on appelle une carte de densité électronique (aujourd'hui numériquement), c'est-à-dire pour chaque point de l'espace évaluer la présence d'électrons en ce point. L'ordinateur détecte les taches, calcule leur intensité, repère leur position et essaie de remonter à la densité électronique pouvant expliquer ces taches.
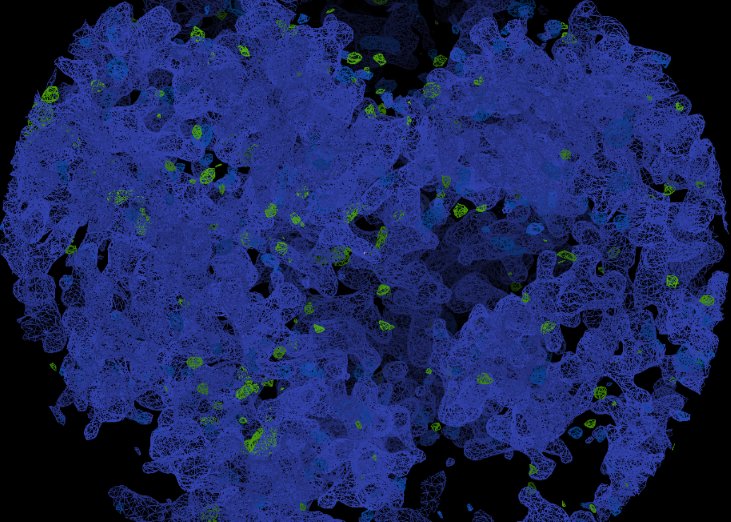
Carte de densité de la transcriptase inverse du VIH.
Si la résolution est suffisamment basse, alors on peut placer les atomes dans ces cartes. On a donc la structure atomique de la protéine.
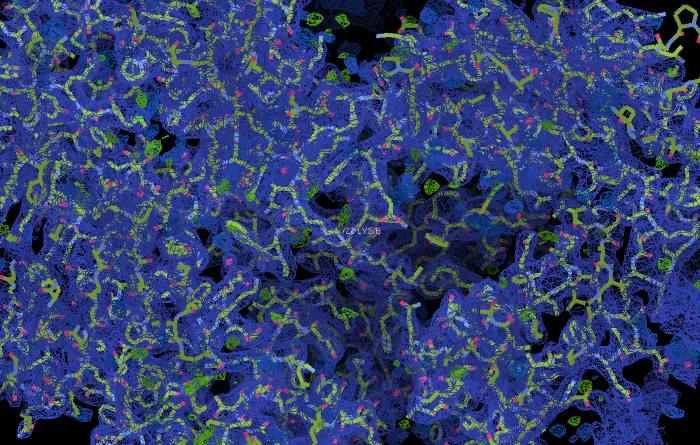
Les atomes ont été positionnés dans la carte de densité.
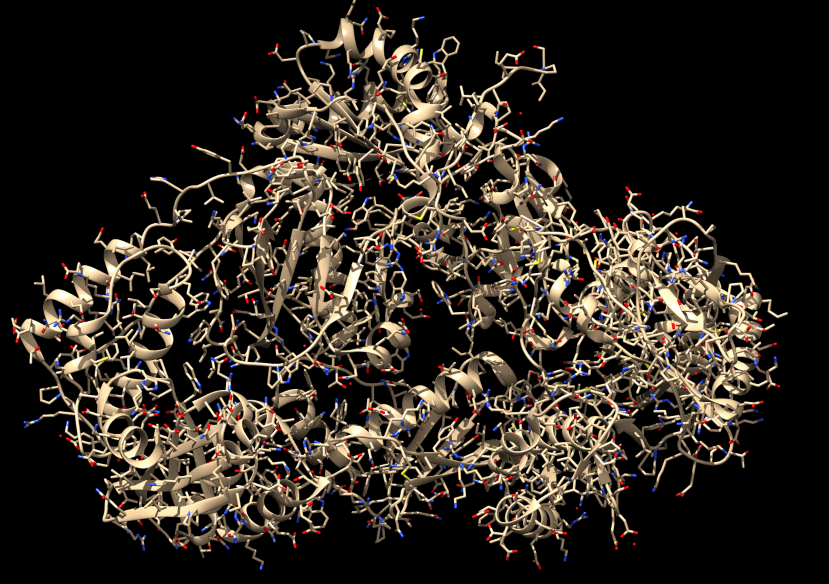
C'est probablement plus visible comme cela…
À la fin, on obtient quelque chose comme ceci. La structure de la protéine est connue très précisément.
Les hélices et les flèches dans la structure atomique sont des conventions de représentation. Ils indiquent que sur la zone de l'hélice, les acides aminés qui s'enchaînent ont une forme en hélice ; pour les flèches, en une structure dite en feuillet. Ce sont deux motifs assez fréquents que l'on représente ainsi pour alléger un peu et faciliter la vue en 3D. À notre niveau, peu importe pour le moment, ce sont juste des conventions d'affichage pour simplifier la vie de ceux qui travaillent sur ce genre d'images.
La Résonance Magnétique Nucléaire ou RMN
C'est une technique plus récente qui exploite la réponse des noyaux certains atomes aux champs magnétiques (résonance, magnétique, nucléaire), principalement des atomes 1H, 13C, 15N, 31P. Explication rapide du phénomène : les noyaux des atomes ont ce qu'on appelle un un moment de spin, ce que l'on peut simplifier et dire que c'est comme une rotation sur lui-même, orienté haut ou bas. Les atomes peuvent changer de spin (ou on les contraint à le faire). En absence de champ magnétique, les deux états sont équiprobables et il n'est pas nécessaire de fournir de l'énergie pour changer de spin.
En présence d'un champ magnétique, le spin orienté dans le sens du champ est favorisé par rapport à l'autre. Il faut alors fournir de l'énergie pour passer d'un spin à un autre, puisque le changement n'est pas favorable. Plus le champ est fort et plus l'un des spins est favorisé par rapport à l'autre donc plus le champ est fort, plus il faut fournir d'énergie.
On force cette transition en émettant des ondes électromagnétiques à la bonne fréquence ; chaque photon contient une énergie $E$ selon la relation de Planck $ E = h \nu $ avec $\nu$ la fréquence de l'onde et $h$ une constante. Il faut que l'énergie du photon apporte exactement l'énergie nécessaire pour la transition ; la transition est ensuite détectée. L'atome résonne alors à la fréquence $\nu$ qu'on a dû mettre pour fournir la bonne énergie.
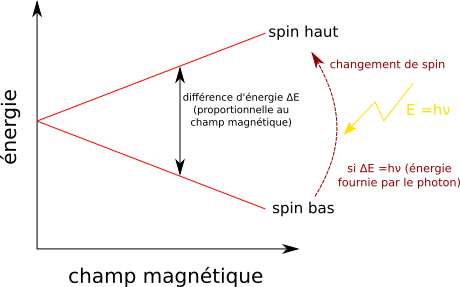
L'atome change de spin s'il reçoit exactement la bonne énergie.
L'intérêt, c'est que la structure de la molécule, sa forme vient perturber localement le champ magnétique ressenti par chaque atome de la molécule. Ainsi, chaque atome ressent un champ magnétique modifié par son voisinage. Donc, la fréquence de résonance est, elle aussi, perturbée par rapport à ce qu'elle devrait être si l'atome était complètement isolé. Ainsi, la fréquence de résonance de chaque atome est porteuse d'une information sur la structure. On scanne tous les atomes pour voir à quelle fréquence ils résonnent. Toujours pareil, calculs informatiques lourds pour déduire les effets de chaque atome sur ses voisins et ainsi dresser la position des atomes.
Il faut des champs magnétiques assez puissants pour que les micro-différences de champ soient détectées. Sinon, elles passeraient inaperçues.
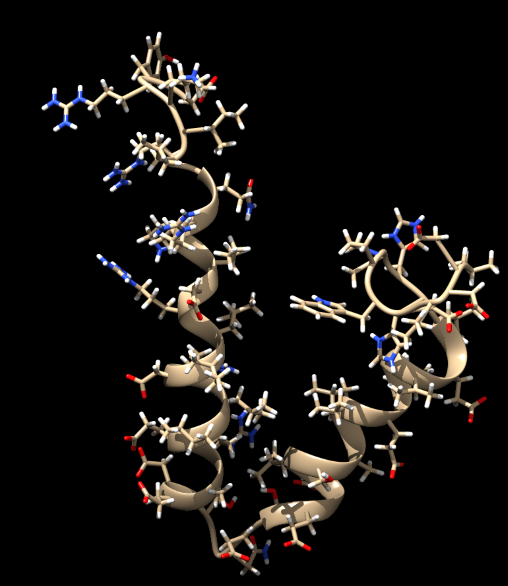
Une protéine accessoire, Vpu.
Chose amusante, ici on peut voir les atomes d'hydrogène (les boules en blanc qui dépassent). Vous pouvez regarder plus haut, il n'y en avait pas avant en cristallographie. C'est possible de les voir aux rayons X mais c'est très rare car il faut une résolution suffisamment fine (globalement moins de 1 Å) pour les voir, c'est-à-dire la distance entre les atomes H et les atomes de carbone, d'oxygène ou d'azote. Très souvent la résolution ne permet pas de conclure sur leur position. On peut essayer de les placer, mais c'est un pari risqué, donc à voir si ça vaut vraiment le coup de les afficher. La RMN voit les atomes H donc on peut les placer sans ces soucis. C'est toujours intéressant de les voir, ils peuvent aussi être impliqués dans des interactions protéine-protéine.
La RMN a ses avantages (inutile de faire cristalliser la protéine, voir les atomes H) mais est assez vite limitée par la taille de la protéine. Plus il y a d'atomes et plus le signal est difficile à isoler. Elle ne se substitue donc pas à la cristallographie - rayons X mais constitue une méthode différente, parallèle.
-
Rappelez-vous, chaque onde électromagnétique se comporte comme une onde (avec sa longueur d'onde) et comme un corpuscule. Le photon est le grain élémentaire de lumière. ↩
Dernière étape : mettre enfin au point le médicament !
On a maintenant en main une structure atomique des protéines impliquées et on compte bien s'en servir. Il faut quelque chose qui épouse assez bien la forme, qui aille se positionner là où ça gêne le plus et qui se fixe de la façon la plus forte possible. Donc il faut :
- repérer sur la structure là où c'est le plus intéressant
- produire un truc de la bonne taille et de la bonne forme
- mettre les groupements chimiques adéquats pour maximiser les interactions
- voir ce que ça donne
- optimiser
- finaliser le produit
Bien entendu, ça prend du temps, des manipulations supplémentaires pour bien identifier la partie à perturber, faire des essais, et ainsi de suite. Mais l'idée est là.
On va présenter l'exemple d'un médicament, l'enfuvirtide. Il va gêner la fusion en perturbant bien comme il faut la protéine gp41 (protéine d'enveloppe du VIH qui intervient dans la fusion avec la molécule, rappelez-vous).
Parfois, on n'arrive pas à avoir la structure atomique de la protéine entière, mais de seulement une partie. C'est mieux que rien et c'est souvent suffisant pour travailler. Ici, sur les images suivantes, on ne verra que des petites parties de gp41.
La protéine gp41 fonctionnelle, lors de l'interaction avec les récepteurs cellulaires, présente une structure à 6 hélices. Cette structure à 6 hélices est nécessaire pour accomplir sa fonction.
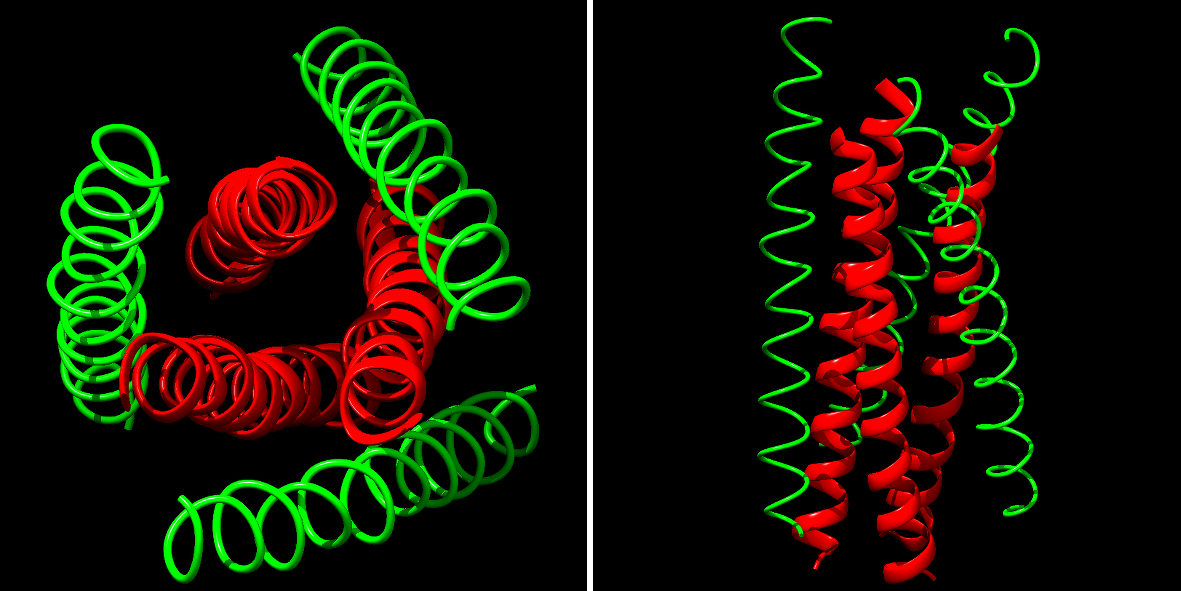
À gauche, vue de dessus. À droite, vue de profil.
Il s'agit de la forme active de gp41 ; au centre, 3 hélices (en rouge), en périphérie, 3 hélices (en vert) également. Nous, on va les repérer en parlant des hélices centrales et des périphériques. Les 3 hélices centrales établissent des interactions entre elles et chaque hélice centrale lie une hélice périphérique. Tous les atomes n'apparaissent pas par souci de clarté de l'image (mais la structure est résolue à l'échelle atomique).
Ici, les 3 hélices centrales sont identiques, de même que les 3 périphériques. La structure est symétrique par rotation de 60° depuis le centre. On a donc finalement 3 dimères de type hélice centrale - hélice périphérique.
Ce qu'il faut, c'est bloquer la formation de la structure à 6 hélices. Le mieux serait de mettre quelque chose qui irait sur une hélice (donc sur ses 2 hélices homologues, par symétrie) et s'y lierait tellement fort que les autres hélices qui devaient se rajouter ne le peuvent pas, la place est déjà occupée.
L'enfuvirtide a ainsi été proposé comme candidat. Il s'agit d'un petit peptide (comme une protéine, mais en plus court) d'environ 30 acides aminés. Voyons ce que ça donne comme interaction :
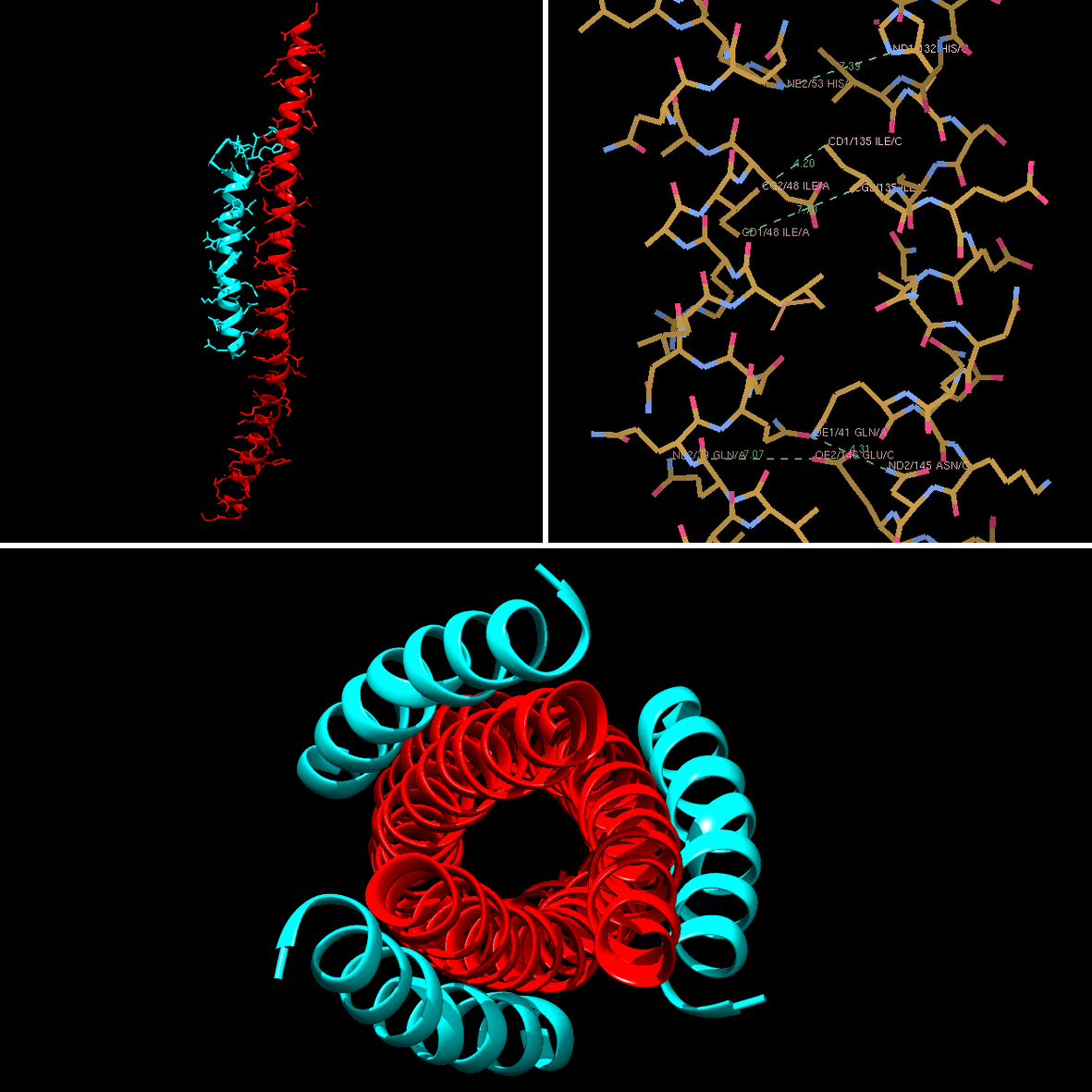
En haut à gauche, l'enfuvirtide (en bleu) et gp41 (la chaîne correspond à une chaîne centrale sur l'image précédente). À droite, un zoom sur les acides aminés entre les deux chaînes. En bas, la structure totale obtenue par interaction gp41 - enfuvirtide.
Puisque les 3 hélices centrales sont symétriques, en mettant au point un inhibiteur se liant à une hélice centrale, il est logique de le voir 3 fois. C'est toujours rassurant de voir que c'est bien le cas !
Cet inhibiteur a une structure en hélice qui vient mimer la forme de la chaîne voisine. Les acides aminés qui le composent ont été choisis pour reproduire cette forme, mais également pour maximiser les interactions au niveau des chaînes latérales. Il vient mimer la forme en hélice donc il peut optimiser les contacts et se coller dessus. Les chaînes latérales des acides aminés stabilisent l'interaction avec gp41 et font de la colle.
Donc, la structure de l'inhibiteur est très adaptée pour se coller sur gp41 et empêcher la formation de la structure à 6 hélices, nécessaire à l'infection de la cellule. Donc, si tout se passe bien, l'infection est bloquée parce qu'un boulet est venu se glisser dans la structure nécessaire à l'infection et bloque l'ensemble de la mécanique. Et il est très difficile à faire partir.
On ne va pas détailler plus que ça les interactions entre l'hélice et l'enfuvirtide, ce serait trop long. On va juste admettre que la molécule a été conçue pour que ça marche bien, au niveau de la forme globale comme au niveau des groupements chimiques pour coller le plus possible à sa cible. Et que ça a l'air de marcher.
Il est hautement probable qu'un premier candidat a été proposé pour venir se fixer, puis qu'on a regardé ce que ça donne et que les interactions ont été optimisées ensuite, en remplaçant certains acides par d'autres pour affiner les interactions. En procédant à tâtons, on peut comme ça optimiser la complémentarité de forme et la complémentarité des acides aminés pour que l'inhibiteur reste le plus possible lié à sa cible.
Bien entendu, il reste à vérifier que le médicament a bel et bien une action positive par rapport à l'infection, par des analyses poussées. On vient de proposer un candidat et de montrer qu'il est pertinent, on n'a pas encore prouvé qu'il avait un rôle. Pour cela, on procède à des essais d'infection de cultures de cellule par le virus, avec et sans la molécule pour comparer les effets. Ensuite, on vérifie que la molécule n'a pas non plus d'effets secondaires sur un organisme entier (et donc un cobaye), cette fois-ci, sur un simple tas de cellules en laboratoire. Finalement, on teste véritablement son activité sur des patients. Si les tests sont concluants, alors on peut enfin dire que le médicament est au point. Dans le cas qu'on vient juste de voir, le produit a bel et bien été validé et autorisé sur le marché.
Voilà, vous savez tout !
C'est une façon de procéder pour mettre au point de nouveaux traitements ; ici contre le VIH, afin de limiter les effets du virus dans le cadre d'une trithérapie, mais le principe reste le même dans de nombreuses autres situations. Globalement, on veut une molécule qui perturbe le plus un virus ou une bactérie pathogène et cette méthode, comme on l'a vu, permet d'arriver à ses fins. Le revers de la médaille, c'est que ça prend du temps, ça avance pas à pas.
On a parlé du fait qu'on pouvait multiplier les cibles en pourrissant plusieurs protéines du virus. C'est le principe de la trithérapie. Le VIH a la fâcheuse tendance de muter fréquemment. Le problème pour nous, c'est d'une part que ça lui permet d'échapper au système immunitaire plus facilement mais surtout que très rapidement, il trouve une parade contre nos pitites molécules. Donc ce qu'on fait, c'est qu'on donne plusieurs traitements à la fois : il est alors beaucoup plus difficile d'échapper aux 3 traitements en même temps. On gagne ainsi du temps avant que le virus ne développe une résistance, mais il risque à tout moment de trouver une parade. C'est pour ça qu'on essaie de trouver le plus de traitements possibles, pour avoir une banque de médicaments suffisamment grande pour toujours avoir des molécules en réserve.
Au passage, on a parlé de cycle viral, de protéines, de structures atomiques, de façon de les obtenir et enfin comment s'en servir. Bien entendu, chaque étape a été simplifiée (impossible de tout décrire facilement et de toute façon ce serait trop pénible) ; en revanche, vous êtes devenus des pros de la synthèse de nouveaux médicaments. Pas trop mal, non ? 
Je tiens à remercier chaleureusement ceux qui m'ont apporté leur aide, lors de l'écriture de cet article, pierre_24 pour toutes ses remarques, et lors de la validation Arius pour tous ses efforts pour corriger et améliorer le texte. Un grand merci à eux 
Sources des images :
- Protein Data Bank (PDB). C'est une base de données de structures protéiques

- logiciels pour obtenir les images : WinCoot et Chimera
- Wikipédia
- Icône d'illustration de l'article : comprimés de paracétamol et d'ibuprofène par ParentingPatch via Wikipédia sous licence CC BY-SA 3.0.
- l'article sur le VIH pour l'image du cycle viral et la page anglaise pour la structure de la particule virale et du génome viral
- l'article sur les fentes d'Young pour la diffraction







